Kurs:Statistik für Anwender/Varianzanalyse
Varianzanalyse
Einfaktorielle Varianzanalyse
Situation und Hypothesenpaar
Situation: Gegeben sind normalverteilte ZV deren Standardabweichungen (bzw. Varianzen) gleich sind.
Hypothesenpaar:
Äquivalent dazu (unter obigen Voraussetzungen):
Benötigte Daten
unabhängige Stichproben
Mittelwerte
Man berechnet aus den Stichproben:
- die Gruppenmittelwerte (’mean of groups’):
- den Gesamtmittelwert (’grand mean’):
Beachte: Es gilt:
Streumaße I
- Die Summe der quadratischen Abweichungen aller Stichprobenwerte vom Gesamtmittelwert bezeichnet man mit:
- Weiterhin bezeichnet man die Summe der quadratischen Abweichungen aller Stichprobenmittelwerte vom Gesamtmittelwert mit:
SST ist ein Maß für die Unterschiede zwischen den verschiedenen Stichproben.
Streumaße II
- Schließlich bezeichnet man die Summe der quadratischen Abweichungen aller Stichprobenwerte vom jeweiligen Stichprobenmittelwerte mit: SSE ist ein Maß für die Unterschiede innerhalb der einzelnen Stichproben.
Man beachte, dass gilt:
Streumaße III
Man teilt nun SST und SSE durch die Zahl der jeweiligen Freiheitsgrade (falls gilt, sind und jeweils -verteilt mit bzw. FG) und erhält die sogenannten ’mittleren quadratischen Abweichungen’
Teststatistik
Aus SST und SSE berechnet man nun die Teststatistik wie folgt:
Idee: MSE stellt eine Schätzung für die Streuung innerhalb der einzelnen Stichproben dar. Im Gegensatz dazu schätzt MST die Streuung der verschiedenen Stichprobenmittelwerte um den Gesamtmittelwert. Nimmt man an, dass gilt, sollte MST (im Vergleich zu MSE) klein sein, folglich ist eine Teststatistik, bei der man eher kleine Werte erwartet, falls gilt.
p-Wert
Zur Berechung wird die F-Verteilung (bzw. Fisher-Verteilung) mit den ’Freiheitsgraden’ und benötigt. Es gilt:
(Dies geht in R mit .)
Durchführung in R
Einlesen der Stichproben in einen gemeinsamen Vektor x zusammen mit einem Faktor g (gleicher Länge), der angibt, zu welcher Größe die jeweiligen Komponenten von x gehören. Dann:
Beispiel 1.1
Verschiedene Drahtsorten () werden auf Zugfestigkeit untersucht. Dabei soll zu geprüft werden, ob die verschiedenen Drahtsorten (oder einige der Sorten) im erwarteten Mittel unterschiedliche Zugfestigkeiten aufweisen. Dazu nimmt man an, dass die ZV , die die Zugfestigkeiten der verschiedenen Sorten beschreiben, normalverteilt mit gleicher Varianz sind und formuliert die Nullhypothese:
Beispiel 1.2
Man erhält folgende Daten (in ):
Daraus berechnet sich:
und
Folglich zeigen die Daten keine siginifikanten Unterschiede zwischen den Zugfestigkeiten der verschiedenen Drahtsorten. Die Nullhypothese ist mit den Daten vereinbar.
Beispiel 1.3
Man kann obige Rechnungen auch in R durchführen lassen. Dies geht etwa mit
Anmerkungen I
- Die oben genannten Voraussetzungen für die Varianzanalyse mit dem F-Test können (und sollten) mit Hilfe von Vortests empirisch geprüft werden. Die Normalverteilungsannahme kann mit Shapiro-Wilks-Tests für jede der ZV getestet werden. Die Annahme der Varianzgleichheit kann man dann mit einem sogenannten Bartlett-Test prüfen. Liefert einer der Vortests ein signifikantes Ergebnis (bzw. einen kleinen -Wert), so kann der F-Test nicht verwendet werden. Man muss dann auf andere Testverfahren zurückgreifen. Beispielsweise kann dann der Test von Kruskal und Wallis verwendet werden.
Anmerkungen II
- Falls die Varianzanalyse ein signifikantes Ergebnis liefert, wird dadurch lediglich angezeigt, dass nicht alle gleich sind. Zur Klärung der Frage,welche der signifikant als verschieden angesehen werden können, stehen weitere Testverfahren zur Verfügung, bespielsweise der Scheffé-Test oder der Tukey-Test.
Aufgabe 1.1
In einer Studie soll untersucht werden, wie sich die Wildschweinpopulationsdichte auf den Traubenertrag im Weinbau auswirkt. Für die Studie wurden 3 Gebiete identifiziert, in denen Weinbau betrieben wird, die jedoch unterschiedliche Populationsdichten an Wildschweinen aufweißen. In jedem Gebiet befinden sich unterschiedlich viele Versuchsflächen, die jeweils gleich bewirtschaftet werden. Bei jeder Testfläche wurde am Ende der Traubensaison der Ertrag an Trauben in Tonnen pro Hektar ermittelt. Es ergeben sich folgende Daten:
Aufgabe 1.2
- Worauf sollte bei der Auswahl der Testgebiete geachtet werden? (Stichwort Randeffekte)
- Überprüfen Sie mit dem Shapiro-Wilks- und dem Bartlett-Test (in R - keine Rechnung notwendig), ob die Voraussetzungen für die Einfaktorielle Varianzanalyse gegeben sind.
Der Tukey-Test (bzw. die Tukey-Methode)
Situation, Signifikanzniveau und Nullhypothese
Situation: Gegeben sind normalverteilte ZV deren Standardabweichungen (bzw. Varianzen) gleich sind.
Signifikanzniveau: Es muss ein Signifikanzniveau festgelegt werden.
Nullhypothesen: für mit
Alle diese Nullhypothesen werden gemeinsam getestet. Das bedeutet: Falls alle wahr sind, erhält man höchstens mit Wahrscheinlichkeit mindestens ein signifikantes Ergebnis.
Benötigte Daten und Teststatistik
benötigte Daten: unabhängige Stichproben gleicher Länge
Die Gesamtlänge ergibt sich dann offenbar als .
Teststatistik: Man berechnet zunächst paarweise die (betragsmäßigen) Differenzen der empirischen Mittelwerte, also
und die mittlere quadratische Abweichung der Fehler
Daraus berechnen sich die Teststatistiken als
Offenbar sprechen hohe Werte dieser Statistik gegen .
p-Werte
-Werte: Zur Berechung wird die studentisierte Spannweite mit den ’Freiheitsgraden’ und benötigt. Zur Nullhypothese ist der -Wert gegeben durch:
(Dies geht in R mit ptukey(x,m,n-m).)
Für die Paare , deren -Wert kleiner oder gleich sind, kann also die entsprechende Nullhypothese verworfen werden. Man hat dann ein oder mehrere signifikante Ergebnisse zum gemeinsamen Niveau , d.h. es wurde berücksichtigt, dass man mehrere Nullhypothesen getestet hat.
Durchführung in R
Einlesen der Stichproben in einen gemeinsamen Vektor x zusammen mit einem Faktor g (gleicher Länge), der angibt, zu welcher Größe die jeweiligen Komponenten von x gehören. Dann:
Zur Ausgabe gehören neben den -Werten der einzelnen Nullhypothesen auch Konfidenzintervalle zum (gemeinsamen) Niveau für die Differenzen der wahren Erwartungswerte . Diese Intervalle lassen sich mit auch graphisch darstellen.
Das bedeutet, dass (falls alle gelten) die Wahrscheinlichkeit, dass alle berechneten Konfidenzintervalle die entsprechende wahre Erwartungswertdifferenz enthalten, mindestens ist.
Variante
Für unterschiedliche Stichprobenlänge von berechnet man die Teststatistiken wie folgt (Tukey-Kramer-Methode):
Beispiel 1.1
Wir betrachten die folgenden Stichproben () zum Signifikantsniveau .
Man berechnet und . Damit ergibt sich der -Wert einer Varianzanalyse mit einem -Tests zur Nullhypothese als . Man kann also davon ausgehen, dass sich Erwartungswerte einiger der Größen unterscheiden.
Beispiel 1.2
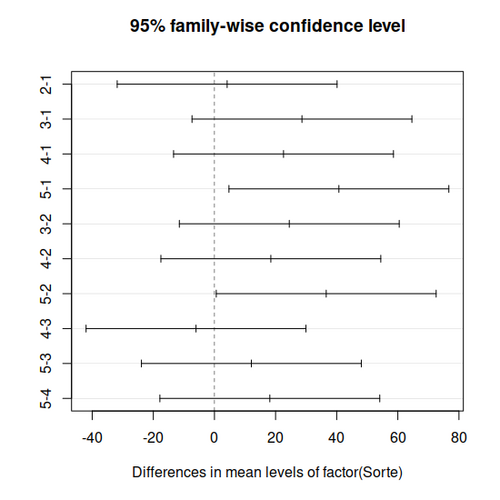
Man möcht nun genauer wissen, welche der Erwartungswerte sich im einzelnen unterscheiden. Dazu führt man einen Tukey-Test durch. Insgesamt werden dabei 10 Vergleiche durchgeführt:
Damit wird also signifikant angezeigt, dass sich die Großen und und auch die Großen und hinsichtlich ihrer Erwartungswerte unterscheiden.
Beispiel 1.3
Legt man ein Signifikanzniveau fest, so kann man mit TukeyHSD eine Graphik erzeugen, die Konfidenzintervalle zum gemeinsamen Niveau zeigt:
Aufgabe
Nehmen Sie nun an, dass Sie in der vorherigen Aufgabe ein Signifikantes Ergebnis erhalten hätten. Berechnen Sie nun mithilfe der Tukey-Methode, zwischen welchen Gruppen signifikante Unterschiede existieren.
Zweifaktorielle Varianzanalyse
Es soll untersucht werden, ob eine ZV (Zielvariable) durch zwei vorliegende Faktoren und beeinflusst wird. Die Faktoren und nehmen dabei nur endlich viele Werte (bzw. Ausprägungen) an ( Möglichkeiten für und Möglichkeiten für ).
Betrachtung als Zufallsvariablen
Man unterscheidet nun ZV
wobei die Größe für die -te Ausprägung von und die -te Ausprägung von ist .
Voraussetzung und Nullhypothese
Vorausgesetzt für den folgenden Test wird, dass alle normalverteilt mit gleicher Varianz sind.
Man untersucht dabei die Nullhypothesen
Stichproben
Zu jeder der vielen Größen benötigt man nun eine Stichprobe
(Wir gehen der Einfachheit halber davon aus, dass alle Stichproben die gleiche Länge haben. Für ungleiche Stichprobenlängen wird es nochmals erheblich komplizierter.)
Zu berechnende Werte und Parameter I
Daraus berechnet man nun die folgenden Mittelwerte und Stichprobenlängen:
- Einzelne Stichprobe ( fest):
Zu berechnende Werte und Parameter II
- Mit festem Wert für zusammengefasste Stichprobe ( fest):
Zu berechnende Werte und Parameter III
- Mit festem Wert für zusammengefasste Stichprobe ( fest):
Zu berechnende Werte und Parameter IV
- Gesamte Stichprobe:
Quadratsummenzerlegung I
Es gilt die folgende Quadratsummenzerlegung:
Quadratsummenzerlegung II
Dabei ist:
Teststatistik
Zu den oben angegebenen Nullhypothesen berechnet man nun Teststatistik und -Wert wie folgt:
Durchführung in R
Man kann diese auch mit R durchführen. Man trägt dazu in einen Vektor x die Daten ein und in zwei Faktoren a und b (beide haben die gleiche Länge wie x) die Information, zu welcher Ausprägung von bzw. die Daten gehören. Dann erhält man mit:
die benötigten Werte (Freiheitsgrade, Quadratsummen, mittlere Quadratsummen, Teststatistiken, -Werte).
Beispiel 1.1
Gegeben seien die Faktoren und mit den Ausprägungen und , dem Signifikanzniveau und der folgenden Stichprobe:
Beispiel 1.2
Es ergibt sich:
Somit kann die Nullhypothese, dass unabhängig von ist, verworfen werden.
Weiterhin ergibt sich:
Somit kann auch diese Nullhypothese, dass unabhängig von ist, verworfen werden.
Weiterhin ergibt sich:
Die Nullhypothese, dass es keine Wechselwirkung zwischen und im Hinblick auf gibt, kann nicht wiederlegt werden, somit ist es anzunehmen, dass es keine Wechselwirkung gibt.
Seiteninformation
Diese Lernresource können Sie als Wiki2Reveal-Foliensatz darstellen.
Wiki2Reveal
Dieser Wiki2Reveal Foliensatz wurde für den Lerneinheit Kurs:Statistik für Anwender' erstellt der Link für die Wiki2Reveal-Folien wurde mit dem Wiki2Reveal-Linkgenerator erstellt.
- Die Seite wurde als Dokumententyp PanDocElectron-SLIDE erstellt.
- Link zur Quelle in Wikiversity: https://de.wikiversity.org/wiki/Kurs:Statistik%20f%C3%BCr%20Anwender/Varianzanalyse
- siehe auch weitere Informationen zu Wiki2Reveal und unter Wiki2Reveal-Linkgenerator.