Kurs:Mathematische Modellbildung/Themen/Modellierung des Infektionsgeschehens durch SARS-CoV-2/Zyklus 3
Das SI-Modell
Ein sehr einfaches Kompartiment-Modell ist die Unterteilung einer Population in lediglich zwei Kompartimente:
- Anteil der Nicht-Infizierten (Susceptible) an der Gesamtbevölkerung N: Eine Zahl zwischen 0 und 1
- Anteil der Infizierten (Infected)
In der Gruppe befinden sich Individuen, welche einmal durch den Erreger infiziert wurden (unabhängig davon, ob sie Symptome zeigen oder nicht).
Susceptibles können von dem Kompartiment durch eine Infektion in das Kompartiment wechseln.
Exponentielles Wachstum
Eine sehr einfache Annahme besteht darin, dass jeder Infizierte Susceptibles (aus einem unendlichen Vorrat) mit einer festen Rate infiziert.
- ,
also exponentielles Wachstum mit einer Infektionskontaktrate und als Anteil an Infizierten am Tag 0.
Das SIR-Modell
Beim vorigen SI-Modell infiziert ein Mitglied von mit einer konstanten Infektionskontaktrate "endlos" weiter.
Dies ist natürlich eine grobe Vereinfachung, der man durch die Einführung der Klasse (Removed) im Kompartiment-Modell abhelfen will.
Das SIR-Modell
Die Erhaltungsgleichung wird um den Anteil erweitert: .
Die Infektionsfunktion übernimmt man unverändert aus dem SI-Modell.
Die Gesundungs-Funktion führt nun Anteile aus nach mit einer Genesungsrate über:
Das SIR-Modell
Beim SIR-Modell kommen also zum einen neue Infizierte hinzu (, genauso wie beim SI-Modell), andere wechseln in die Gruppe ():
Das SIR-Modell
amit haben wir ein kleines System von Differentialgleichungen mit
Wegen ergibt sich .
Damit ergibt sich die dritte Differentialgleichung auch aus . Sie braucht daher nicht integriert zu werden.
Lösung des Gleichungssystems mit Maxima
Auch Differentialgleichungssysteme lassen sich mit der rk()-Funktion leicht lösen. In den Klammern werden lediglich mehrere Gleichungen hintereinander eingefügt und durch Kommata getrennt:
/*SIR-Modell*/
beta: 0.3219;
gamma: 0.1;
I0: 1.93E-07;
sol: rk([ -beta*S*I ,
beta*S*I-gamma*I],
[S,I],
[(1-I0),I0],
[t,0,160,1])$
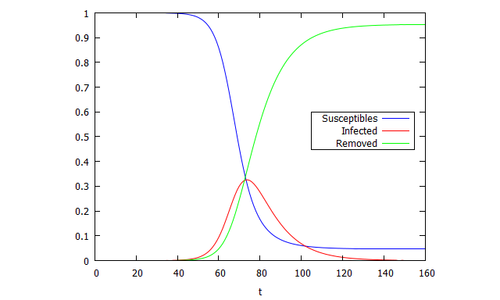
plot2d ([[discrete,makelist([p[1],p[2]],p,sol)],
[discrete,makelist([p[1],p[3]],p,sol)],
[discrete,makelist([p[1],1-p[3]-p[2]],p,sol)]] ,
[xlabel,"t"],
[legend,"Susceptibles","Infected","Removed"],
[color, blue, red,green],
[gnuplot_preamble, "set key box at 155.,.6" ])$
Die Basisreproduktionszahl
Eine oft genannte Kenngröße bei Epidemien ist die Basisreproduktionszahl . Sie gibt an, wie viele Andere eine Infizierte Person während der Phase der Infektiosität durchschnittlich ansteckt.
Für das SIR-Modell gilt :
- .
Mit den Werten für und würden wir als erhalten. Dies liegt innerhalb der Schätzwerte .