Kurs:Mathematische Modellbildung/Themen/Corona-Modellierung/Einführung Teilprojekt 3
Zur Navigation springen
Zur Suche springen
Thema
- Vergleich der Infektionsverläufe des Covid-19 Virus in 4 ausgewählten Ländern
- SIR Modell
- dort getroffenen Maßnahmen zur Bekämpfung
Begründung der Länderauswahl
- Deutschland: Identifikation durch uns selbst
- Italien : sehr früh und sehr stark betroffen
- Schweden : meisten Maßnahmen waren freiwillig
- Südkorea : geringe Fallzahlen
SIR-Modell
- konstant
- Interaktion innerhalb der Gruppen mit gleicher Wahrscheinlichkeit
- -> ->
- Infizierte sind sofort ansteckend
Anfangsbedingungen:
- R0=0
- I0 >0, S0 >0
- α > β
Differentialgleichungen des SIR-Modells
- , mit α: Infektionsrate
- , mit β: Sterberate
Modellierung
Sekundarstufe I
Erstellen des SIR-Modells für die ausgewählten Länder mithilfe der Tabellenkalkulation
SIR-Modell TK
Modellerstellung mithilfe eines Tabellenkalkulationsprogramms[3] mit
- Zeit + Zeitschritt
- Infizierbare Personen
- Infizierte über "next I": Nächstes It: Wenn < 1 und * BRZ* >0, dann * BRZt* . Sonst 0
- Genesene Personen
- BRZ frei wählbar
Vorgehen
- 1. Recherchieren der realen Fallzahlen und Verläufe in dem jeweiligen Land
- 2. Anpassung von BRZ, bis modellierte Kurven mit den realen Kurven übereinstimmen
- 3. Modellieren, wie sich Verläufe verändern, wenn sich ab bestimmten Zeitpunkten die BRZ erhöhen/verringern
SIR - Schweden
SIR - Südkorea
SIR - Italien
SIR - Deutschland
Sekundarstufe II
- Verteilung des Corona-Virus zwischen den Ländern
- Integrierung einer Transportmatrix in das SIR-Modell
Grundlagen Matrizenrechnung
- lineares Gleichungssystem der Form
- Matrizenmultiplikation nur wenn im Falle und
- Beispielhafte Erklärung: Matrix und Vektor
Anwendung auf Flugverkehr
- : Anteil der in Land i Bleibenden [%]
- : Anteil der Reisenden von Land i nach Land j [%]
- Vektor : Einwohnerzahlen von Land i und Land j
- Vektor : Einwohnerzahlen der Länder nach Transportprozess
Abschätzung, wie viele Flugpassagiere von einem Land zum anderen fliegen:
- Annahme =
- Berechnung mit abfliegenden Personen pro Tag weltweit und abfliegenden Personen pro Tag des Landes
- Flüge unabhängig von der Entfernung der Länder und der Zeit
Beispiel: Transportprozesse zwischen Schweden und Südkorea
und lassen sich analog bestimmen
Matrixeinbindung in LibreOffice
"next I" SW: * BRZ * - * ( - )
Infektionsgeschehen in Südkorea und Schweden im Transportmodell
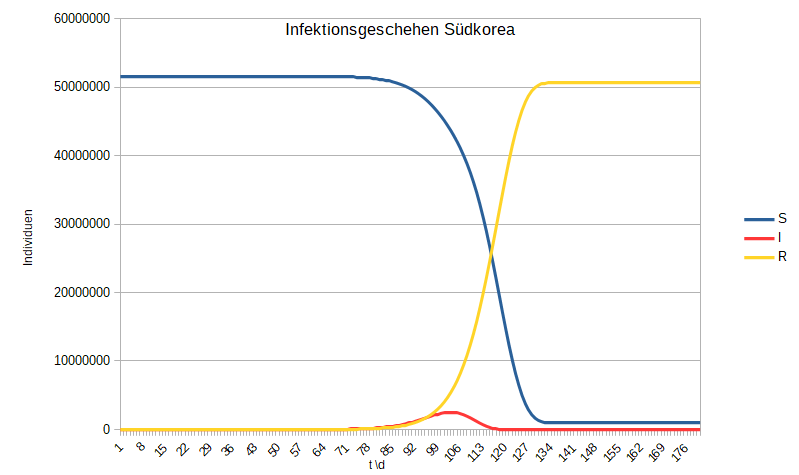
- Erste Infizierte Person in Schweden an Tag 46 (+/- 3 Tage)
- Infektionsgeschehen in Schweden schneller (15 Tage weniger bis Maximum)

Uniniveau
Konzept
- Ziel: Funktion finden, die die Neuinfektionen abbildet
- Logistisches Wachstum über Bernoulli-DGL
- Graph der Lösung der B.-DGL entspricht Kurve der kummulierten Infiziertenzahl
- Ableitung der Lösung entspricht der Kurve der Neuinfektionen
- nichtlineare Regression in R Studio
- Vergleich der Länder über ausgegebene Parameter
Bernoullische Differentialgleichung
Einschub: Bernoullische Differentialgleichung
- Eine DGL der Gestalt lässt sich wie folgt lösen:
Lösung der Bernoullische Differentialgleichung
Lösen der homogenen DGL
- ergibt
- mit die partikulare Lösung ist
- und mit ergibt sich:
- Da
Parametisierung
- Durch Parametisierung
- Beidseitige Ableitung der Bernoullische DGL:
ist eine Lösung der DGL .
SIR: DGL
SIR-Modell
- Für das SIR-Modell werden drei DGL verwendet ():
Es gilt
Lösung der DGL nach Näherung
Die DGL für die Infiziertenzahl lässt sich also wie folgt schreiben:
- Vernachlässigung des letzten Terms :
vergleiche mit ; mit aus der Ableitung der B.-DGL
Modellierung der Infiziertenzahl
- nicht lineare Regression mit der Methode der kleinsten Quadrate
- Kurvenanpassung an (bspw. ) Werte durch Minimerung des Abstandquadrates der Messpunkte zu der Fitkurve über die Veränderung der Parameter:
Minimum finden über
- Schätzintervall für die Werte von und (Werte liegen zu darin)
- : Schwere der Epidemie
- : Ausbreitungsgeschwindigkeit
- : Dauer zum vorläufigen Höhepunkt der Epidemie
Ergebnis

- Schwere der Pandemie in Italien am höchsten, in Südkorea am geringsten
- Südkorea höchste Ausbreitungsgeschwindigkeit
- Schweden geringste Ausbreitungsgeschwindigkeit
- Italien: Peak der Neuinfektionen am frühsten (Tag 35)
- Schweden Peak am spätesten (Tag 53)
Beispiel: Deutschland

Optimierung
- Verbesserte Anpassung der Kurve durch die Ableitung der Gompertz-Funktion (asymmetrische Sigmoidfunktion)
- Parameter analog zur vorherigen Funktion, nur durch ersetzt
- [Geogebra: Parametereinstellungen der Fitfunktion]
Ergebnis

- Ergebnisse ähnlich wie in Tabelle davor
- Schwedens Peak wird 4 Tage früher erreicht
- Ausbreitungsgeschwindigkeit Italiens unterscheidet sich deutlicher von der Schwedens
Beispiel: Deutschland

Vergleich

Siehe auch
- [[../Vergleich der Länder/|Vergleich der Länder - Theoretische Informationen]]
- SIR-Modell
Seiteninformation
Dieser Wiki2Reveal Foliensatz wurde für den Lerneinheit Kurs:Mathematische Modellbildung' erstellt der Link für die Wiki2Reveal-Folien wurde mit dem Wiki2Reveal-Linkgenerator erstellt.
- Die Seite wurde als Dokumententyp PanDocElectron-SLIDE erstellt.
- Link zur Quelle in Wikiversity: https://de.wikiversity.org/wiki/Kurs:Mathematische%20Modellbildung/Themen/Corona-Modellierung/Einf%C3%BChrung%20Teilprojekt%203
- siehe auch weitere Informationen zu Wiki2Reveal und unter Wiki2Reveal-Linkgenerator.



